Contact Press / Media
Dr. Marius Spohn
Stellv. Abteilungsleiter »Naturstoffforschung«
Fraunhofer-Institut für Molekularbiologie und Angewandte Oekologie IME
Ohlebergsweg 12
35392 Gießen
Telefon +49 641 97219-189
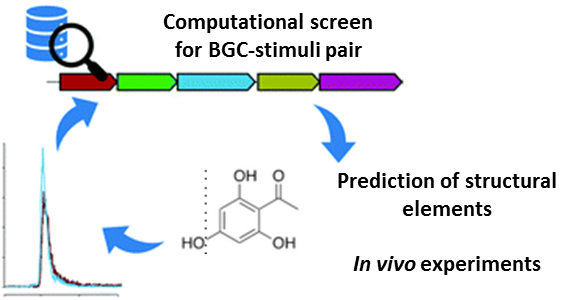
Mikrobielle Naturstoffe (NPs) waren schon immer eine grundlegende Quelle der Inspiration für die Entwicklung vieler, uns heute dienlicher Antiinfektiva und Schädlingsbekämpfungsmitteln. Diese Stoffe werden als nicht essentiell für das Wachstum und die Reproduktion von Mikroorganismen angesehen. Sie sind eher ein Adaptationsmechanismus, der unter sehr spezifischen Umweltbedingungen einen evolutionären Fitnessvorteil bedeutet. Folglich sind die Biosynthesegene der Naturstoffe nicht konstitutiv exprimiert und unter Standard-Laborbedinungen kann oft keine Produktion detektiert werden. Dieses Phänomen ist eine grundlegende Herausforderung in Kultivierungs-abhängigen, sowie –unabhängigen NP Discovery-Projekten. Die Notwendigkeit eines adäquaten Stimulus, der die NP-Biosynthese induziert zu finden, bedingte eine Vielzahl an Strategien hervor, die beispielsweise darauf abzielen die geeignete Umweltbedingung im Labor zu imitieren. Bei Erreichen eines erfolgreichen Versuchsaufbaus führt das schlussendlich zur Expression der abgeschalteten Biosynthesegene und in Konsequenz zur Produktion der Naturstoffe von Interesse.
Während einem Erasmus+ Praktikums in unserer Naturstoffabteilung durchsuchte M.Sc. Isabella Tropiano bakterielle Genome in silico nach bestimmten Sequenzmotiven, die es ermöglichen einen gegebenen Umweltstimulus mit einem Biosynthesegencluster in Verbindung zu bringen. Diese Genomstudie bedingte die Auffindung einer bisher unbekannten Familie von Gencluster, die unter typischen Fermentationsbedinungen abgeschaltet bleiben. Lediglich unter einem künstlich hervorgerufenen Mangel eines spezifischen Spurenelements konnten die identifizierten Produzentenstämme angeregt werden die Zielsubstanzen zu produzieren. In der kurzen Zeit des Praktikums gelang es einen eleganten Prozessablauf zu entwickeln, der genomische Vorhersagen als grundlegendes Element einbezieht um die NP-Produktionsprofile gezielt zu manipulieren. In Kombination mit unserer Metabolomics-Plattform konnten die gefundenen Biosynthesecluster mit einer außerordentlichen Diversität von bekannten aber auch unbekannten Naturstoffen korreliert werden.
| Projekttitel | From in silico prediction of environmental stimulus via hypothesis driven experimental design to awakening dormant gene clusters |
|---|---|
| Laufzeit | 11/2020 –06/2021 |
| Förderung | Erasmus+ |
| Projektleiter | Dr. Marius Spohn |
| Ziele | In silico
In vivo
|