Molekulares Umweltnebenwirkungsscreening


Die Wirkstoffe aus Pflanzenschutzmitteln, Bioziden, Arzneimitteln oder Kosmetika gelangen entweder gezielt oder über ihre Anwendung am Menschen oder an Materialien in die Umwelt und können dort eine schädliche Wirkung auf Nicht-Zielorganismen ausüben. Die Testung der Umweltwirkung einer stetig wachsenden Zahl an neu entwickelten Wirkstoffen stellt einen immer größer werdenden Kostenfaktor für die Wirkstoff-produzierende Industrie dar. Darüber hinaus sind die regulatorisch geforderten OECD-Tests zeit- und ressourcenaufwändig und somit nicht kompatibel mit einem Screening von Substanzvorläufern.
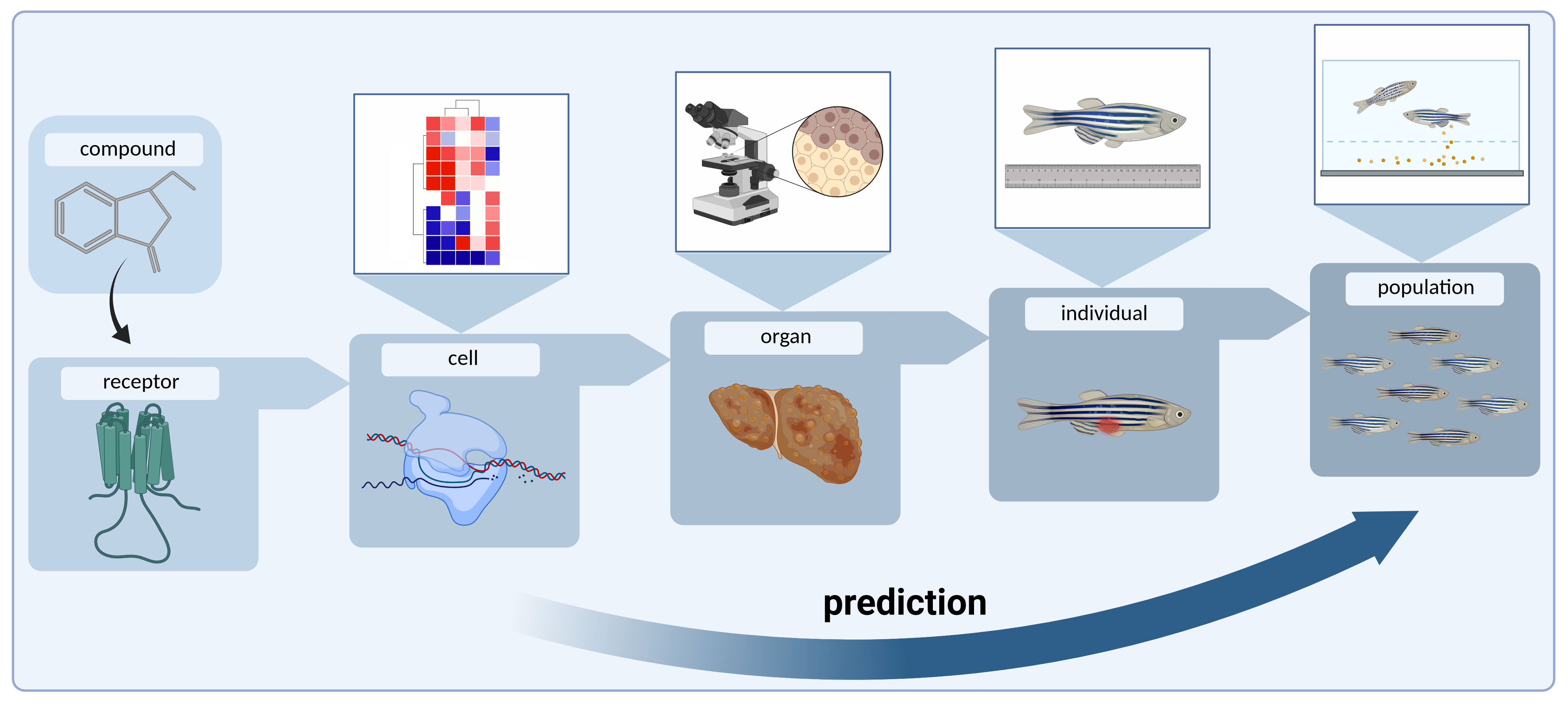
Die Anwendung modernster OMICs-Methoden ermöglicht eine sensitive und gleichzeitig genomweite Identifizierung von Substanz-induzierten molekularen Veränderungen in ökologisch relevanten Organismen auf der Ebene der DNA (Epigenom), der RNA (Transkriptom) und der Proteine (Proteom). Das Wissen über die von einem Stoff hervorgerufenen molekularen Veränderungen und deren direkte Verknüpfung mit Phänotypen und Populationseffekten eröffnet die Möglichkeit einer frühzeitigen Identifizierung ökotoxischer Substanzen und erleichtert die Klassifizierung von Stoffen in Bezug auf ihre Umweltwirkung. Darüber hinaus können solche systembiologischen Untersuchungen mit minimalen Mengen an Bioressourcen durchgeführt werden, was sie zu einem vielversprechenden Ansatz zur frühen Erfassung der Umweltwirkung von Stoffen im Rahmen eines Screenings macht. Gleichzeitig ermöglichen sie eine signifikante Reduzierung der Anzahl an Tierversuchen.
Die OMICs-basierte Identifizierung solcher Stoffwechselwege, welche durch eine Substanz gestört werden, trägt wesentlich zur Etablierung von Adverse Outcome Pathways (AOPs) bei. Das AOP-Konzept dient zur unmittelbaren Verknüpfung initialer molekularer Ereignisse mit den daraus folgenden schädlichen Effekten für den Organismus und die Population und erfährt auch in der behördlichen Stoffregulation zunehmende Aufmerksamkeit.
Die Abteilung Ecotoxicogenomics kombiniert ökotoxikologische Richtlinientests mit OMICs-Methoden (RNA-Seq, quantitative LC-MS/MS), um frühe molekulare Veränderungen zu erfassen, welche schädlichen Effekten auf den Organismus und die Population vorhergehen. Unsere Datenbank molekularer Fingerabdrücke wird in Screening-Methoden zur Umweltrisikovorhersage von Substanzvorläufern genutzt. Die Verfügbarkeit eines solchen Umweltnebenwirkungsscreenings während der industriellen Wirkstoffentwicklung vermeidet die kostspielige Weiterentwicklung von Vorläufersubstanzen mit einem hohen ökotoxischen Potenzial und ermöglicht eine nachhaltige Entwicklung von umweltsicheren Wirkstoffen.