Die klassische Pflanzenzüchtung zu beschleunigen und zu verbessern wird unter dem Gesichtspunkt einer stetig steigenden Weltbevölkerung immer wichtiger. Der Mangel an kultivierbaren Anbauflächen und das vermehrte Auftreten von Ernteausfällen aufgrund globaler Wetterextreme erfordert die immer schnellere Entwicklung neuer und verbesserter Getreidepflanzen zur Minimierung zukünftiger Nahrungsengpässe. Um dabei transgene Ansätze vermeiden zu können besteht ein hoher Bedarf an innovativen analytischen Techniken, die es erlauben die genetischen Eigenschaften junger Keimlinge zu identifizieren und gleichzeitig Rückschlüsse bzgl. des Auftretens bestimmter verbesserter phänotypischer Merkmale zu ziehen.
Derzeit zum Einsatz kommende molekulare Züchtungsansätze wie „Selection with Markers and Advanced Reproductive Technologies“ (SMART) oder „Marker Assisted Selection“ (MAS) haben grundsätzlich das Potenzial die Pflanzenzüchtung in gewissem Umfang zu verbessern, sind aber auch mit Nachteilen verbunden, die einen Durchbruch bei der Generierung von Pflanzen mit all den gewünschten Eigenschaften bisher verhindern.
Unser Ziel am Fraunhofer IME ist daher die Entwicklung neuartiger technologischer Ansätze, die eine schnelle Hochdurchsatzanalyse von Pflanzenmerkmalen auf molekularer Ebene ermöglichen, und so zu einer Vereinfachung und deutlichen Beschleunigung des Selektionsprozess während der Züchtung beitragen.
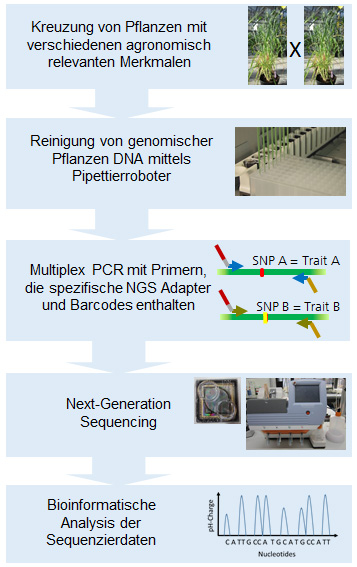
Bisher entwickelt wurden beispielsweise ein Multiplex-PCR Ansatz zur Zygositätsbestimmung und Analyse der Allelverteilung bei Mais sowie ein innovativer Sequenzieransatz auf Basis von Next-Generation-Sequencing (NGS) zur Hochdurchsatz-Selektion von Pflanzen auf züchtungsrelevante Merkmale. Wir konnten für wichtige Getreidepflanzen wie Gerste oder Mais demonstrieren, dass das Next-Generation Sequencing von kleinen PCR Produkten, die bestimmte Polymorphismen beinhalten oder flankieren, als effektives Werkzeug bei der Selektion von Pflanzen im Rahmen des Züchtungsprozesses eingesetzt werden kann. So konnten wir zeigen, dass die Sequenzierung sehr schnelle und verlässliche Aussagen bzgl. der vorliegenden Genotypen und der jeweiligen Allelhäufigkeit (Zygosität) ermöglicht. Diese Methode ist daher ideal geeignet um große Mengen von Pflanzen parallel zu untersuchen und Vorhersagen bzgl. des späteren Phänotyps zu treffen, da bis zu 80 Millionen individuelle Reads in einem einzigen Sequenzierlauf generiert werden können. Damit lassen sich hunderte von Proben verschiedener Pflanzenlinien bzw. genomischer Merkmale poolen und parallel analysieren. Für Pflanzenzüchter sind diese neuen Erkenntnisse von großer Relevanz, da sie i.d.R. sehr große Populationen auf die verschiedensten Merkmale hin selektieren müssen. Unser Next-Generation-Sequencing Ansatz erlaubt es nun schnell und kostengünstig mittels Genotyping spezifischer Polymorphismen bzw. genomischer Merkmale auf den späteren Phänotyp zu schließen. Das Analyseverfahren kann auch bei anderen ökonomisch bedeutende Kulturpflanzen eingesetzt werden und insgesamt zu einer signifikant schnelleren und zielgerichteteren Pflanzenzüchtung beitragen.
 Fraunhofer-Institut für Molekularbiologie und Angewandte Oekologie IME
Fraunhofer-Institut für Molekularbiologie und Angewandte Oekologie IME