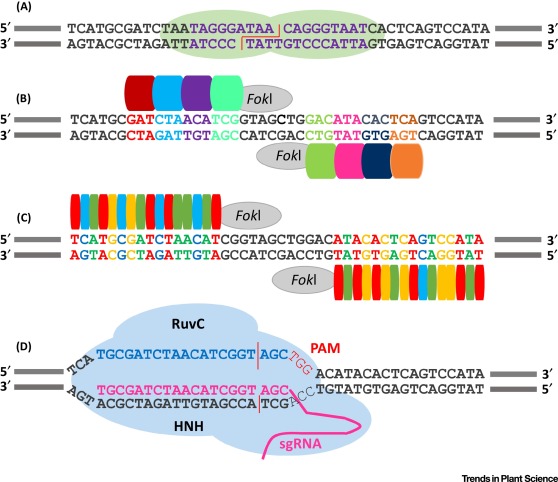
Sequenzspezifische Nukleasen werden genutzt, um DNA Doppelstrangbrüche an ausgewählten Stellen im Pflanzengenom zu erzeugen. Dadurch werden an diesen Stellen DNA Reparaturmechanismen induziert, die für unterschiedliche Zwecke genutzt werden können. Neben der gezielten Abschaltung unerwünschter Genfunktionen, können insbesondere auch Genoptimierungen erreicht werden. Die Technologie erlaubt weiterhin das gezielte Einbringen von Transgenen an definierten Stellen im Pflanzengenom. Dadurch können Modell- und Nutzpflanzen mit veränderten Eigenschaften erzeugt werden.
In unserer Abteilung nutzen wir sowohl Zinkfingernukleasen als auch das CRISPR/Cas9 System, um gezielte Veränderung im Genom von Pflanzen und Pflanzenzellkulturen zu erzeugen. Gemeinsam mit unseren Partnern aus Industrie und Wissenschaft entwickeln wir Pflanzen mit verbesserten Eigenschaften und arbeiten an der stetigen Optimierung der eingesetzten Verfahren in Modellorganismen, um optimale Ergebnisse zu erzielen.
 Fraunhofer-Institut für Molekularbiologie und Angewandte Oekologie IME
Fraunhofer-Institut für Molekularbiologie und Angewandte Oekologie IME